單細胞測序技術指的是通過高通量測序技術(NGS,Next Generation Sequencing)分析每一個單個細胞的序列信息,從而更高分辨率地揭示細胞間的細胞差異以及其在微環境中的功能情況。
爲什麽要做單細胞測序
單細胞測序發展的十余年,使得我們所認知的這個世界越來越精准。我們傳統的測序方法,是在組織水平或者說多細胞水平上進行的,最終得到的數據其實是從多個或者多類細胞中得到的平均值,但是無法明確細胞異質性的信息。而單細胞測序,則能夠在更精細的單個細胞水平上獲得遺傳信息,從而揭示每個細胞的基因結構和基因表達狀態,反映常規測序所無法得到的異質性信息和稀有細胞的遺傳信息。因此也有人稱單細胞測序爲NGS的“顯微鏡”。
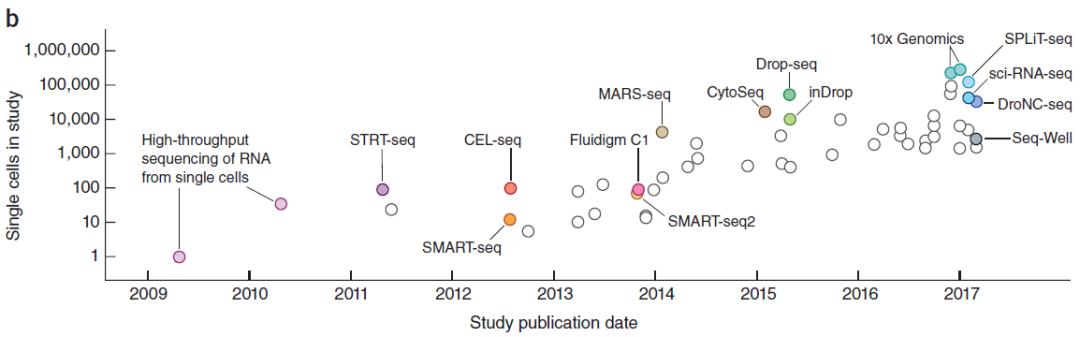
圖1. 單細胞測序發展曆程
單細胞測序技術的難點
單細胞測序最大的局限性在于待測核酸的含量,如一個細胞裏的DNA或RNA含量僅僅處在皮克 (picograms) 級水平,遠遠達不到現有建庫試劑盒的最低投入量需求。因此,必須先對單細胞內的微量核酸分子進行擴增,而且必須保證盡可能少地出現技術誤差,以便開展後續的測序及其它研究。其次高通量單細胞測序需要增加捕獲效率,提高靈敏度。除此之外,樣本處理過程中對于稀有樣本的獲取和難解離組織的細胞分離,也是很大的挑戰。
主要的幾種單細胞測序技術
◎ 基于微流控液滴的10× Genomics
10×genomics 公司的 Chromium 系統,利用微流控技術將細胞自動分配到 100,000到 1,000,000微反應體系,每個微反應體系含有一種特定的 barcode 序列;含有 barcode 信息的凝膠珠子(GEMs)首先與樣品和酶的混合物混合,然後與位于微流體「雙十字」連接中的油表面活性劑溶液結合;GEMs 流到儲液器中並進行收集。凝膠珠子溶解釋放 barcode 序列,開始對樣本進行標記;最後將每個液滴中含有 barcode 信息的産物混合,然後構建標准測序文庫。
10×genomics的優勢在于細胞通量高,建庫周期短,操作簡便,具有超高的捕獲效率。不足之處在于只能獲得3’端的轉錄本信息,且樣本要求高。
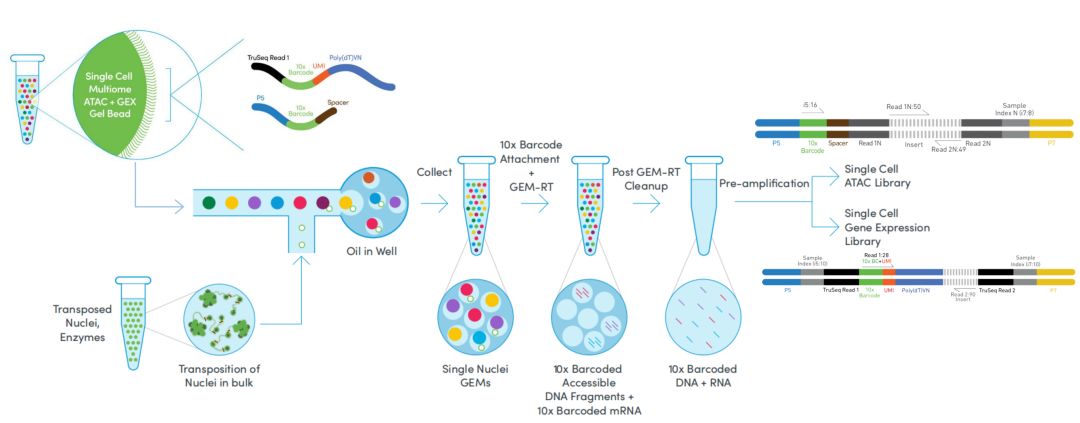
圖2. 10×genomics測序原理
◎ 基于微孔板的BD Rhapsody
BD Rhapsody 技術利用卡式芯片在磁性的寡核苷酸條形碼標記微球上實現單細胞捕獲和 mRNA 轉錄本的分子標簽,然後將這些微球合並到單個管中用于 cDNA 擴增和文庫構建。可滿足 100~10,000 個細胞的自動分選、擴增及建庫。此外,該技術使用帶有寡核苷酸的高質量抗體(Ab-oligos),這條寡核苷酸帶有抗體特異的條形碼,細胞在經過 Ab-oligos 標記後,可在單細胞水平同時獲得轉錄組和蛋白表達。
相比于10×genomics,BD Rhapsody在捕獲效率和有效性上有一定優勢。

圖3. BD Rhapsody測序原理
◎ 基于PCR擴增的Smart-seq技術
Smart-Seq(Switching mechanism at 5’ end of the RNA transcript)是一項具裏程碑意義的技術。細胞被裂解後,將RNA與包含oligo(dT)的引物雜交。然後添加幾個無模板的C核苷酸,生成第一條鏈。這種poly(C)垂懸只添加到全長轉錄本上。然後將寡核苷酸引物與poly(C)突出雜交,合成第二條鏈。全長cDNA經過PCR擴增,以獲得納克級的DNA。PCR産物純化後可用于測序。在後來又有學者對該技術進行改進,新方案使用鎖核酸(LNA)、更高濃度的MgCl2以及甜菜堿。它不再需要純化步驟,可大大提高産量。
Smart-seq相比較于10×genomics檢測到的轉錄本更多,但是不足在于測序reads無法實現鏈特異性,同時由于對聚腺苷酸化的RNA具有選擇性,因此不適用于分析非poly(A)的RNA。
圖4. Smart-seq測序原理
◎ 基于線性擴增的MARS-seq技術
通過 T7 啓動子連在 oligo dT 引物上,可以在 cDNA 合成後啓動 IVT(in vitro transcription ),MARS-seq 2.0 是在 MARS-seq 基礎上,整合 index 分選以及大量平行單細胞 RNA測序方法,從而形成的一套流程。
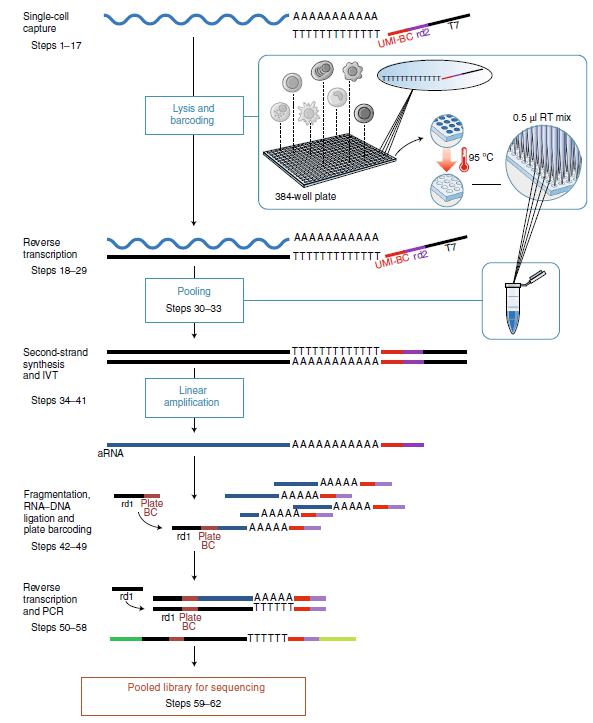
圖5. MARS-seq測序原理
單細胞測序技術發展前景
2013年,單細胞測序技術被《Nature Methods》評爲年度技術。同年,《Science》將單細胞測序列爲年度最值得關注的六大領域榜首。同時隨著技術發展更新,單細胞測序技術由早期的高成本、低通量及低自動化發展到目前的低成本、高通量、高自動化,臨床應用得到飛速發展,能夠快速確定成千上萬個細胞的精確基因表達模式,分析每個細胞的遺傳異質性。單細胞測序技術發展至今僅10余年,在神經生物學、腫瘤研究、産前基因診斷等多個領域發揮重要作用,早已成爲科學研究領域當之無愧的焦點。
參考文獻
<下拉查看>
【1】. Ziegenhain C, Vieth B, Parekh S, et al. Comparative analysis of single-cell RNA sequencing methods[J]. Molecular cell, 2017, 65(4): 631-643. e4.
【2】. Method of the Year 2013. [J]. Nat Methods ,2014,11:1.
【3】. The biology of genomes. Single-cell sequencing tackles basic and biomedical questions.[J] .Science, 2012, 336: 976-7.
【4】. Navin Nicholas E, Delineating cancer evolution with single-cell sequencing.[J] .Sci Transl Med, 2015, 7: 296fs29.
【5】. Svensson Valentine,Vento-Tormo Roser,Teichmann Sarah A, Exponential scaling of single-cell RNA-seq in the past decade.[J] .Nat Protoc, 2018, 13: 599-604.
【6】. Picelli S, Faridani OR, Björklund AK, Winberg G, Sagasser S, Sandberg R. Full-length RNA-seq from single cells using Smart-seq2. Nat Protoc. 2014 Jan;9(1):171-81. doi: 10.1038/nprot.2014.006. Epub 2014 Jan 2. PMID: 24385147.
【7】. Keren-Shaul H, Kenigsberg E, Jaitin DA, David E, Paul F, Tanay A, Amit I. MARS-seq2.0: an experimental and analytical pipeline for indexed sorting combined with single-cell RNA sequencing. Nat Protoc. 2019 Jun;14(6):1841-1862. doi: 10.1038/s41596-019-0164-4. Epub 2019 May 17. PMID: 31101904.
 圖1. 單細胞測序發展曆程
圖1. 單細胞測序發展曆程 圖2. 10×genomics測序原理
圖2. 10×genomics測序原理 圖3. BD Rhapsody測序原理
圖3. BD Rhapsody測序原理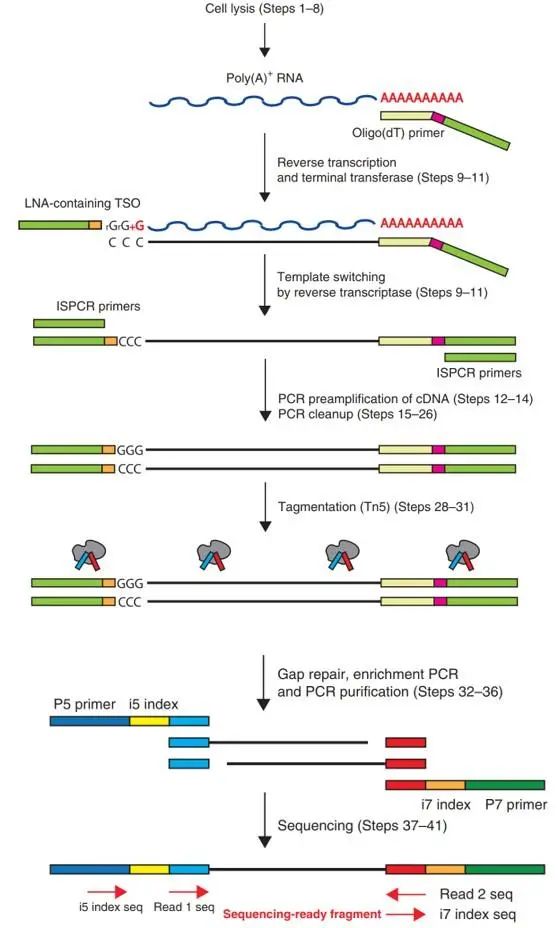
 圖5. MARS-seq測序原理
圖5. MARS-seq測序原理